Komplexe Tabelle
Verfasst: Sa 11. Apr 2015, 09:39
Hallo liebe LaTex Freunde!
Ich verzweifle gerade an einer Tabelle.
Im Anhang ist die Tabelle als Bild, wie sie aussehen soll und auch dann meine Ausgabe.
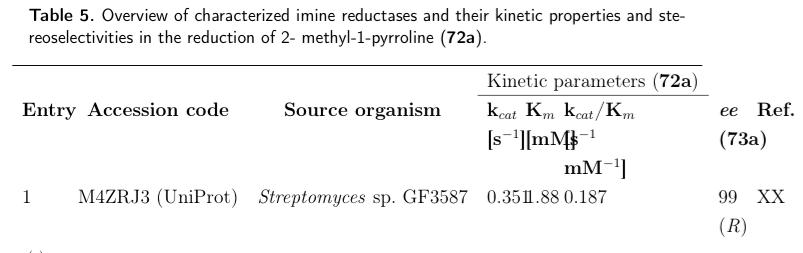
Zielausgabe:
Hier ist mein Quellcode, vl kann mir wer ja sagen, wie ich in TexShop die Tabelle so hinbekomme, dass sie so aussieht wie die Vorlage.
Hab auch schon mit kleinere Schrift probiert, aber die \tabularx Umgebung lässt mich nur den gesamten Text kleiner machen und nicht nur den Inhalt der einzelnen Zeilen.
Vielen Dank schon mal für die Hilfe!
Ich verzweifle gerade an einer Tabelle.
Im Anhang ist die Tabelle als Bild, wie sie aussehen soll und auch dann meine Ausgabe.

Zielausgabe:

Hier ist mein Quellcode, vl kann mir wer ja sagen, wie ich in TexShop die Tabelle so hinbekomme, dass sie so aussieht wie die Vorlage.
Hab auch schon mit kleinere Schrift probiert, aber die \tabularx Umgebung lässt mich nur den gesamten Text kleiner machen und nicht nur den Inhalt der einzelnen Zeilen.
\documentclass[12pt, a4paper]{article}
\usepackage[english, ngerman]{betababel}
\usepackage[T1]{fontenc}
\usepackage[utf8]{inputenc}
\usepackage{tabularx}
\usepackage{threeparttable}
\begin{document}
\begin{table}[H]
\centering
\caption{Overview of characterized imine reductases and their kinetic properties and stereoselectivities in the reduction of 2-
methyl-1-pyrroline (\textbf{72a})}
\begin{threeparttable}
\begin{tabularx}{\textwidth}{p{0.8cm} c c X X p{2.1cm} X X} \hline
& & & \multicolumn{3}{c}{Kinetic parameters (\textbf{72a})} & & \\
\cline{4-6}
\textbf{Entry} & \textbf{Accession code}& \textbf{Source organism}& \textbf{k$_{cat}$ [s$^{-1}$]} & \textbf{K$_{m}$ [mM]} &\textbf{k$_{cat}$/K$_m$ [s$^{-1}$ mM$^{-1}$]} & \textbf{\textit{ee} (73a)} & \textbf{Ref.}\\
1 & M4ZRJ3 (UniProt) & \textit{Streptomyces} sp. GF3587 & 0.351 & 1.88 & 0.187 & 99 (\textit{R}) & XX\\
\end{tabularx}
\end{threeparttable}
\label{tab:Tab5}
\end{table}